胚胎著床是包括人類在內的哺乳動物發育過程中的裏程碑事件💇🏻,生理狀態下超過半數以上的人類胚胎由於無法順利著床導致不孕🎤。既往研究通常使用小鼠和食蟹猴等模式生物對這一過程展開探索🧑🏽⚖️,然而調控圍著床時期胚胎發育的分子機製和形態學變化特征在不同物種之間存在較大差異🦓,這使得在小鼠等模式生物研究中獲得的調控規律較難為人類胚胎發育研究提供有價值的線索🤾🏽。然而✖️,由於人類胚胎著床發生在受精卵形成後一周左右的時間點,這使得研究人員無法獲得生理狀況下這一發育階段的人類胚胎👲🏽。長期以來,這一人類關鍵發育階段一直是發育生物學研究的黑匣子。
為了深入探討這一過程的分子動態規律,挖掘調控胚胎著床過程的潛在分子機製,8月22日🔖,意昂官网生命科學學院、北京未來基因診斷高精尖創新中心(ICG)、生物醫學前沿創新中心(BIOPIC)湯富酬教授課題組攜手意昂官网第三醫院喬傑教授課題組🚍,在Nature在線發表了題為“Reconstituting the transcriptome and DNA methylome landscapes of human implantation”的論文。結合體外模擬人類著床策略1和高精度單細胞多組學測序技術2,3(single-cell RNA-seq, single-cell Trio-seq2)🙆🏿♀️,首次利用單細胞轉錄組和DNA甲基化組圖譜重構了人類胚胎著床過程,系統解析了這一關鍵發育過程的基因表達調控網絡和DNA甲基化動態變化過程✍🏽。研究者發現,圍著床時期人類胚胎的三個主要細胞譜系(上胚層、原始內胚層🫶🏼、滋養外胚層)表現出獨特的發育特征,而且胚胎在這一時期迅速呈現出母胎連接預備狀態;他們發現在可觀察事件窗口期內(體外培養day12以前),雌雄胚胎的X染色體劑量並未達到平衡,且雌性胚胎逐漸啟動並逐漸呈現出父源或母源X染色體隨機失活趨勢;研究者利用單細胞多組學測序手段發現不同細胞譜系具有截然不同的DNA甲基化動態變化特征🩵👰🏼,提示基因表達調控網絡和DNA甲基化可能共同協調決定囊胚階段後的細胞譜系命運。
人類胚胎發育從具有全能性的受精卵開始,伴隨著多次卵裂和分裂從而形成一個具有特定結構的囊胚。囊胚主要由外圍的外滋養層細胞(trophectoderm,TE)和內細胞團(inner cell mass🦤,ICM)細胞組成,成熟的內細胞團會進一步發育成為具有多能性的上胚層(epiblast,EPI)和原始內胚層(primitive endoderm🫳🏽🙍🏼♂️,PE)👨🏿🌾。囊胚著床之後TE主要分化為胚外滋養層結構,例如胎盤等👸;而上胚層主要會發育形成胎體的各個組織器官🦜。隨著體外受精技術的革新,人類早期著床前胚胎發育過程被廣泛研究4,5。然而,在發育的第五至七天,胚胎必須通過著床進入母體子宮壁才能繼續存活和發育,隨後逐漸形成原腸胚🌭,並進一步分化形成各類器官和組織原基🪷。
著床失敗是導致早期流產的重要因素之一6💁🏽。然而研究者們幾乎無法獲得自然受孕後的早期著床後階段的人類胚胎,導致人們對胚胎發育過程中這一關鍵階段的譜系特化和分子調控等規律知之甚少👩🍼。猴子胚胎曾被用於早期胚胎發育研究7,但著床後早期胚胎的猴子模型與人類仍然存在一定的物種差異。小鼠作為最常見模式生物被廣泛應用於早期胚胎研究。人類囊胚與子宮內膜細胞共培養體系為模擬體內著床後發育過程提供了另一實驗策略8👨🏻🍳,然而這類研究策略對於著床後發育過程的重現程度和可重復性存在一定問題🙋♂️。尤其是該體系必須有母體組織的參與,因此較難被推廣使用💆🏿♂️,且無法應用於胚胎的自主性構建(self-organization)研究。2016年,Magdalena Zernicka-Goetz實驗室建立了小鼠著床後胚胎的體外培養體系,該課題組利用獨特的培養條件將小鼠著床前的囊胚進行體外培養,直至形成類似於著床後胚胎的三胚層結構,為闡明早期著床後胚胎發育提供了重要實驗手段9🧖🏻♂️。
不同哺乳動物的胚胎類型存在較明顯的物種差異🛐,因此從小鼠到人類發育過程的研究推理從而獲得的有效結論非常有限🚮,這意味著人類著床後胚胎形態學和分子調控等變化規律最好直接通過人類胚胎研究獲取。相比小鼠,人類囊胚形成過程中的分子和細胞學機製研究仍然偏少🐱,已有研究暗示二者在譜系分化和功能方面存在時間和潛能上的巨大差異10。Ali H. Brivanlou實驗室以及Magdalena Zernicka-Goetz實驗室近期將小鼠著床後胚胎培養體系進一步發展至人類胚胎,新建的培養體系同樣能促進人類胚胎在體外經歷著床前向著床後的轉化與發育,培養體系無需母體組織參與🦹🏽;形態學證據揭示了這一階段人類胚胎發育的主要特征🎛,表明人類胚胎在著床後期具有自我構建(self-organization)特性👴🏿,這極大地豐富了人們對於人類胚胎著床這一生物學過程的認識1,11。
細胞身份識別和命運取決於其基因表達網絡🤵🏻,同時該網絡受到復雜的遺傳和表觀遺傳學等機製調控。解析人類胚胎細胞基因表達的時空特異性模式對於了解胚胎早期發育過程至關重要🦾。將單細胞分辨率組學檢測技術應用於人類胚胎發育研究使人們精確認識這一過程成為可能🧖🏿🚏。湯富酬課題組和喬傑課題組長期緊密合作🗜,致力於包括著床前胚胎在內的人類生殖系細胞發育過程中基因表達、表觀遺傳學調控特征和潛在的機製研究:2013年,結合湯富酬課題組發展的單細胞轉錄組測序技術12,繪製了人類著床前胚胎的高精度單細胞基因表達特征圖譜(Nature Structural & Molecular Biology,2013)13🤾🏻;2014年,該課題組利用新發展的微量細胞DNA甲基化組測序技術系統解析了人類著床前胚胎發育過程中DNA甲基化組重編程動態特征(Nature,2014)14🙏🏼;2018年,該團隊利用單細胞DNA甲基化高通量測序技術在單細胞水平解析了人類著床前胚胎發育的DNA甲基化組圖譜(Nature Genetics,2018)15。同年🔀,他們利用單細胞多組學測序技術解析了人類著床前胚胎發育過程中DNA甲基化組和染色質狀態組的重編程過程,以及染色質狀態與DNA甲基化之間的相互關系(Nature Cell Biology,2018)16。以上研究極大豐富了人們對於人類著床前胚胎基因表達和表觀遺傳調控規律的認知。此外,多個課題組曾利用一系列單細胞技術對人類著床前胚胎的分子表達譜進行系統性解析17,18。然而由於技術限製,胚胎著床這一關鍵發育階段的多維度分子表達特征,尤其是該過程中譜系特異性分子表達譜和潛在的調控規律長期以來一直是未解之謎。
湯富酬課題組和喬傑課題組再次攜手,結合人類胚胎體外培養體系與單細胞組學技術(單細胞轉錄組和多組學測序),系統揭示了人類早期著床後胚胎發育過程中的分子表達圖譜💱,並在此基礎上挖掘了基因表達調控網絡、DNA甲基化動態特征等潛在分子調控機製。
1. 人類胚胎在囊胚階段後期具備體外自我生長能力(體外模擬胚胎著床)
為了證實胚胎在體外無母體組織參與情況下的著床過程💆🏽♀️,研究者根據已報道胚胎培養策略重現了胚胎形態學動態變化特征1,11👄。證據顯示胚胎在無母體組織參與下能發育至第12/14天,胚胎逐漸呈現出特異性的形態學特征,例如滋養外胚層細胞數量大幅度增加,上胚層逐漸形成羊膜腔以及原始內胚層逐漸生長並逐漸包圍上胚層(圖1&視頻)。根據國際倫理學準則🚣🏿♀️,研究者在第14天終止了體外胚胎培養實驗⛹🏽。

圖1 人類胚胎體外模擬著床生長過程(day6-12)
2. 不同細胞譜系的關鍵發育特征以及特征性基因挖掘
研究者利用已知的譜系標誌基因對圍著床時期的胚胎細胞進行譜系鑒定☞,結果顯示胚胎基本維持了囊胚晚期的三個主要細胞譜系(上胚層🛄、原始內胚層、滋養外胚層)。有趣的是👨🏽🦳,各個譜系均逐漸呈現出各自獨特的基因表達特征,例如上胚層呈現出明確的多能性轉變(pluripotency transition),原始內胚層逐漸開始表達卵黃囊發育相關基因(例如CD44),滋養外胚層則逐漸開始表達荷爾蒙相關基因(例如CGB家族基因)🍰。以上特征均提示胚胎在這一關鍵發育階段逐漸呈現出母胎連接預備狀態(圖2)。不同細胞譜系之間的基因表達對比分析,各類細胞譜系均具備獨特的特征性基因(signature gene)👮🏽♀️。且部分基因與已發表食蟹猴圍著床時期胚胎相關譜系的基因表達特征類似(圖3)。

圖2 人類圍著床時期胚胎的譜系鑒定和各自的發育學特征

圖3 人類與食蟹猴著床後胚胎細胞的基因表達圖譜對比分析
OTX2曾被報道是圍著床時期胚胎上胚層(EPI)多能性轉化(pluripotency transition)的重要標誌基因19🕵️♂️,然而本研究的轉錄組數據顯示,OTX2主要表達在原始內胚層(PE),並非在上胚層(EPI)。進一步的全胚胎免疫熒光染色顯示,OTX2表達在部分原始內胚層細胞中🕺,與轉錄組分析結果一致。這一結果表明🧑🏽🚀,具有相同發育來源(內細胞團起源)的兩類細胞(上胚層細胞和原始內胚層細胞)可能攜帶特殊基因表達痕跡(圖4)🤲🏼。這些數據提示該研究中轉錄組數據的潛在資源價值🥧,新的譜系標記基因鑒定可能有助於早期胚胎中的譜系鑒定和幹/祖細胞衍生研究。
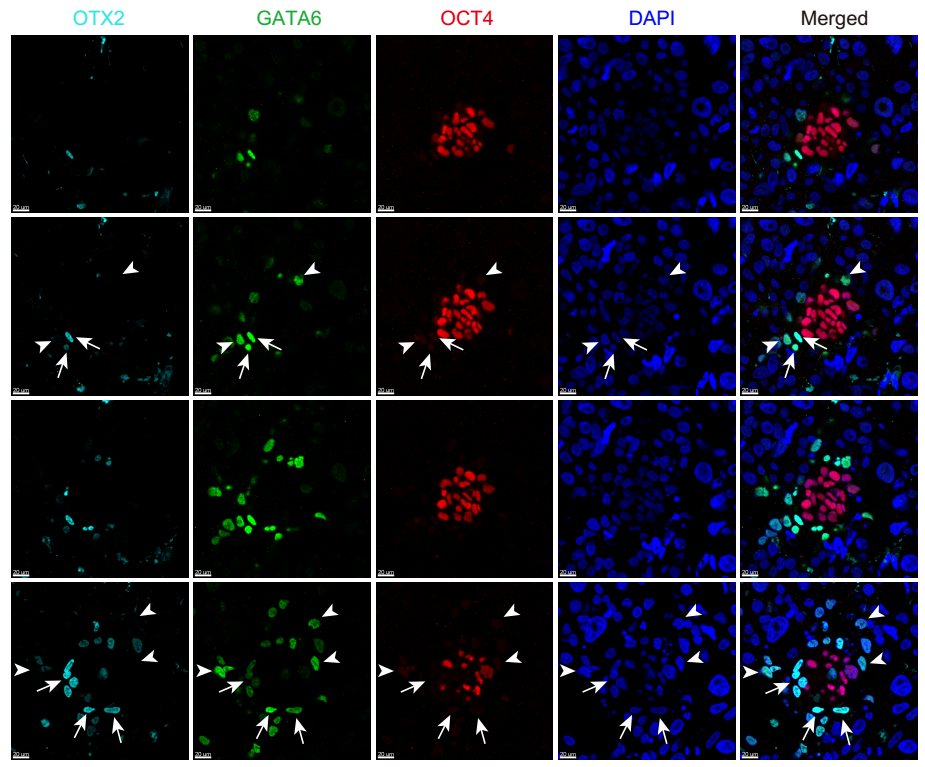
圖4 OTX2在圍著床時期胚胎的表達情況
3. 滋養外胚層特化成為兩類亞群
研究者發現,隨著圍著床時期胚胎的發育🍍,滋養外胚層逐漸形成兩類亞群,其中一類亞群主要表達女性妊娠相關基因,而另一類亞群則幾乎不表達該類基因🏑。這兩類細胞隨後被鑒定為細胞滋養層細胞(cytotrophoblasts🧑🏽🚒,CTs)和合胞滋養層細胞(syncytiotrophoblasts👱🏼,STs)。除激素相關基因外,合胞滋養層細胞(STs)還表達部分新發現基因,例如TCL6和TBX3(圖5)。
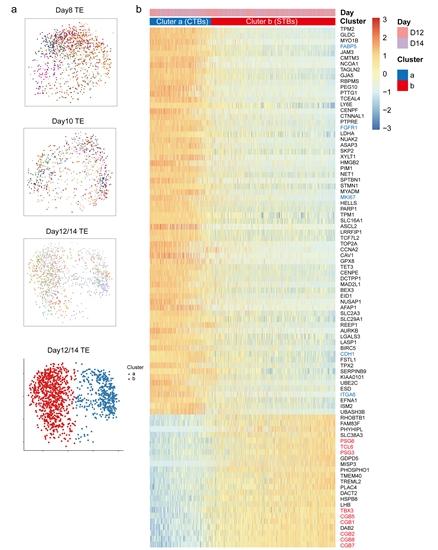
圖5 滋養外胚層(TE)兩類亞群的特征性基因表達譜
4. X染色體劑量平衡
自1961年Lyon等人報道雌雄個體之間的X染色體劑量特征後,X染色體劑量平衡一直是發育生物學關註的焦點之一20。X染色體失活(XCI)對於女性(XX)與男性(XY)之間X連鎖基因的劑量平衡具有重要意義🏃♂️➡️,同時X染色體上調(XCU)對於X連鎖基因與常染色體基因之間的劑量平衡具有重要意義。2016年,Fredrik Lanner團隊曾報道雌雄胚胎的X染色體劑量在囊胚階段幾乎已達平衡🌨,且為X染色體阻滯模式(X-dampening,雌性細胞兩條X染色體劑量均部分下調)17🥡💆♀️。研究團隊捕獲到多個階段的雌雄胚胎,這為系統揭示這一過程中的關鍵生物學細節提供了可能。可追蹤父母源等位基因表達的單細胞全長轉錄組測序分析顯示,在可觀察事件窗口內(day12以前),雌雄胚胎的X染色體劑量並未達到平衡,且雌性胚胎逐漸啟動並逐漸呈現出父源或者母源X染色體隨機失活趨勢(XCI)(圖6)🥷🏻。另一方面,X染色體劑量與常染色體基因的表達量平衡需要通過雌性與雄性的X染色體上調(XCU)來實現,晚期胚胎單細胞中活躍的X染色體(雄性細胞中僅含一條X染色體且處於活躍狀態👩👦🫸,雌性細胞則含有一條活躍的X染色體和一條失活的X染色體)需要上調表達劑量兩倍,以達到跟同一個細胞中每個常染色體兩個拷貝同樣的表達劑量(X染色體/常染色體的表達劑量比從1⏲:2上調至2:2)💒。該研究發現X染色體上調在著床階段的雌性和雄性胚胎細胞均已啟動🫐,但還未達到上調兩倍表達劑量的完成狀態🤸🏽♀️。此外🥉,研究者發現圍著床時期胚胎還存在一定程度的拷貝數變異(CNV),且拷貝數變異並未影響主要譜系的整體基因表達特征。
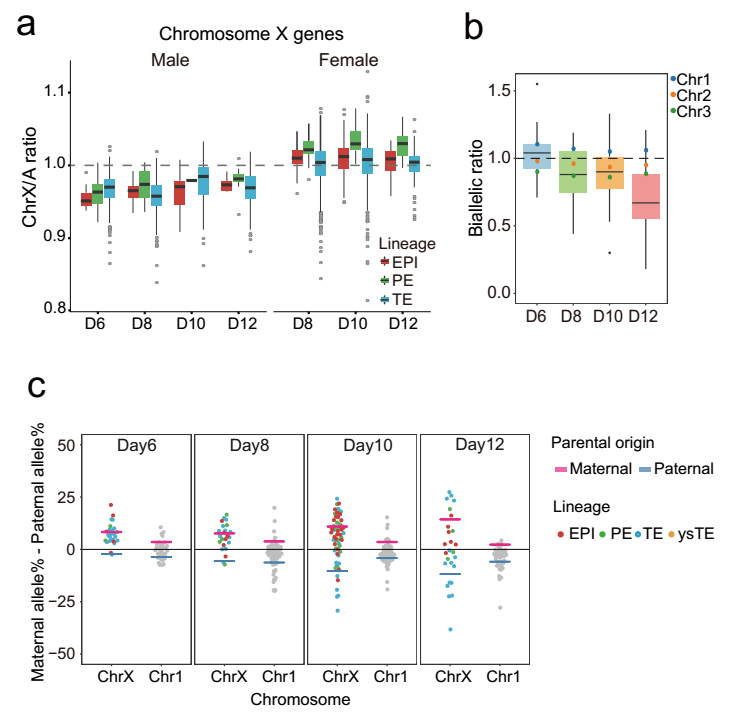
圖6 圍著床時期胚胎的X染色體劑量動態變化過程
5. 譜系特異性DNA甲基化動態變化規律
為進一步分析胚胎著床過程中的基因組甲基化特征☂️,研究人員利用該團隊創建的單細胞多組學測序技術對三類細胞譜系的基因組甲基化過程進行了深度分析。圍著床階段胚胎細胞在DNA甲基化特征水平分布成四個主要細胞群體(圖7):囊胚期(day 6)上胚層/原始內胚層/滋養外胚層、著床後時期(day8/10)上胚層、著床後時期(day8-12)原始內胚層👱🏼♀️、著床後時期(day8-12)滋養外胚層。該結果說明三類細胞譜系在囊胚發育階段(著床前)具有相似的DNA甲基化模式🟠,著床後迅速獲得各自獨特的DNA甲基化特征。
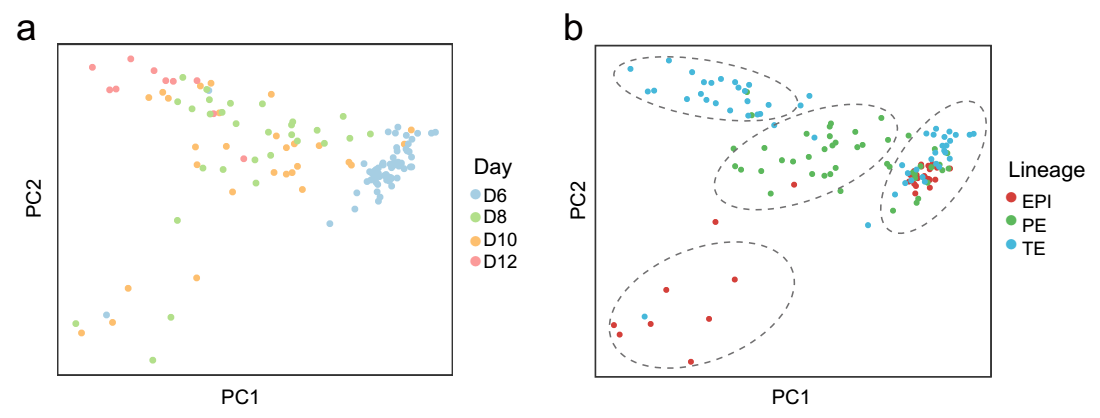
圖7 各細胞譜系在著床過程具有特異性DNA甲基化特征(基於PCA分析)
研究者重點關註三個細胞譜系各自的DNA甲基化動力學變化過程🥿。總體來說🙅🏻♂️🙍🏻♀️,上胚層、原始內胚層和滋養外胚層在囊胚發育階段後基因組均發生了重新甲基化(圖8),表明胚胎著床過程可能經歷了表觀遺傳調控的自我構建過程。其中上胚層的DNA甲基化水平從囊胚的26.1%(day6)顯著增加到著床後胚胎的60.0%(day10)。同樣,滋養外胚層群體的DNA甲基化水平也在囊胚階段(day6)從23.5%上升到著床後胚胎(day10)的46.3%🧍。滋養外胚層群體的DNA甲基化水平顯著低於上胚層譜系(day10),這意味著胚內(上胚層)和胚外(滋養外胚層)譜系細胞的表觀遺傳調控過程可能存在較大差異🍧。出乎意料的是,囊胚期原始內胚層的DNA甲基化水平(27.0%)與上胚層相當(26.1%),第八天上升至33.2%(上胚層為49.9%)👨🏼🚀,第十天略有上升至36.8% (上胚層為60.0%)。與上胚層相比🐌,來自內細胞團的另一個譜系原始內胚層在著床過程中意外地呈現出非常緩慢的DNA甲基化動力學特征(圖8)。這些數據表明,著床後早期胚胎在囊胚期後很快起始了大規模DNA甲基化過程💇🏽♀️,三個主要譜系不但表現出不同的基因表達特征🧑,而且在DNA重甲基化特征呈現不同步的明顯差異。這表明DNA甲基化可能在維持特定細胞譜系的發育過程中發揮重要作用。因此,這暗示著床過程中的調控各個細胞譜系發育的表觀遺傳學特征也存在差異。
有趣的是👨👨👧👧,不同譜系間差異DNA甲基化基因分析表明,各個譜系在胚胎第十天都攜帶著特異性DNA甲基化🕧。例如,多能性基因POU5F1和NANOG在第八天滋養外胚層細胞中被特異性甲基化,而上胚層和原始內胚層細胞則沒有🚃。相反🖕🤙🏻,滋養外胚層發育基因如MMP26和PSG7在上胚層和原始內胚層細胞中特異性甲基化🧜🏿,但在第八天滋養外胚層細胞中卻維持著低甲基化的狀態21,22📬。相比之下,第八天原始內胚層標記基因如APOA1和CPN1在上胚層和滋養外胚層細胞中特異性甲基化,而在原始內胚層細胞中沒有被甲基化(圖7)23,24。這些結果表明,DNA甲基化在著床後早期的時間窗口內可能參與特異性調控了關鍵基因的轉錄👨🏿🌾,與基因轉錄共同協調決定了不同細胞譜系的發育潛能特化過程👨🏿🔬。
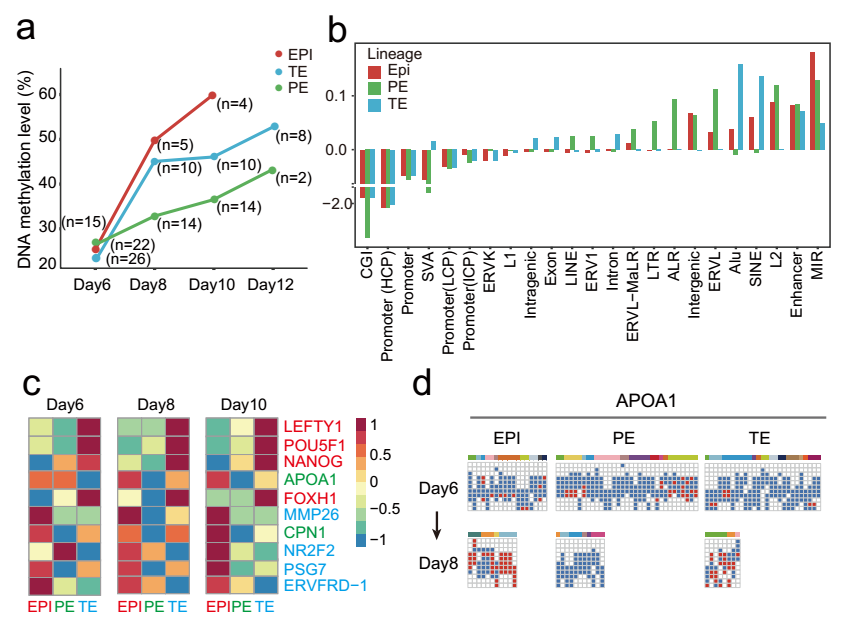
圖8 三類主要譜系在圍著床發育時期的DNA甲基化水平動態變化過程
綜上,這項研究提供的單細胞分辨率的轉錄組和DNA甲基化組數據對於研究早期人類胚胎發育具有潛在重要價值。盡管仍不清楚體內和體外胚胎發育之間存在的具體差異,但該研究提供的數據資源可能為優化模擬體外著床策略提供直接線索與幫助☘️👷🏼♂️,這也為多能幹細胞的分化與發育相關研究提供重要線索和依據。
意昂官网生命科學學院博士後周帆博士🧑🏽🔬、博士生汪睿🤷🏼♂️🖖🏻、博士生毛雨諾和北醫三院博士生袁鵬、博士生任一昕為該論文的並列第一作者,湯富酬教授和喬傑為共同通訊作者。該課題得到意昂官网生命科學儀器中心(成像平臺)和高精尖中心高通量測序平臺的協助與支持👨🏻🦰。
參考文獻
1 Deglincerti, A. et al. Self-organization of the in vitro attached human embryo. Nature533, 251-254, doi:10.1038/nature17948 (2016).
2 Li, L. et al. Single-Cell RNA-Seq Analysis Maps Development of Human Germline Cells and Gonadal Niche Interactions. Cell stem cell20, 858-873 e854, doi:10.1016/j.stem.2017.03.007 (2017).
3 Bian, S. et al. Single-cell multiomics sequencing and analyses of human colorectal cancer. Science362, 1060-1063, doi:10.1126/science.aao3791 (2018).
4 Edwards, R. G., Bavister, B. D. & Steptoe, P. C. Early stages of fertilization in vitro of human oocytes matured in vitro. Nature221, 632-635 (1969).
5 Edwards, R. G., Steptoe, P. C. & Purdy, J. M. Fertilization and cleavage in vitro of preovulator human oocytes. Nature227, 1307-1309 (1970).
6 Koot, Y. E., Teklenburg, G., Salker, M. S., Brosens, J. J. & Macklon, N. S. Molecular aspects of implantation failure. Biochimica et biophysica acta1822, 1943-1950, doi:10.1016/j.bbadis.2012.05.017 (2012).
7 Nakamura, T. et al.A developmental coordinate of pluripotency among mice, monkeys and humans. Nature537, 57-62, doi:10.1038/nature19096 (2016).
8 Weimar, C. H., Post Uiterweer, E. D., Teklenburg, G., Heijnen, C. J. & Macklon, N. S. In-vitro model systems for the study of human embryo-endometrium interactions. Reproductive biomedicine online27, 461-476, doi:10.1016/j.rbmo.2013.08.002 (2013).
9 Bedzhov, I., Leung, C. Y., Bialecka, M. & Zernicka-Goetz, M. In vitro culture of mouse blastocysts beyond the implantation stages. Nature protocols9, 2732-2739, doi:10.1038/nprot.2014.186 (2014).
10 Rossant, J. Mouse and human blastocyst-derived stem cells: vive les differences. Development142, 9-12, doi:10.1242/dev.115451 (2015).
11 Shahbazi, M. N. et al. Self-organization of the human embryo in the absence of maternal tissues. Nature cell biology18, 700-708, doi:10.1038/ncb3347 (2016).
12 Tang, F. et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature methods6, 377-382, doi:10.1038/nmeth.1315 (2009).
13 Yan, L. et al. Single-cell RNA-Seq profiling of human preimplantation embryos and embryonic stem cells. Nature structural & molecular biology20, 1131-1139, doi:10.1038/nsmb.2660 (2013).
14 Guo, H. et al. The DNA methylation landscape of human early embryos. Nature511, 606-610, doi:10.1038/nature13544 (2014).
15 Zhu, P. et al. Single-cell DNA methylome sequencing of human preimplantation embryos. Nature genetics50, 12-19, doi:10.1038/s41588-017-0007-6 (2018).
16 Li, L. et al. Single-cell multi-omics sequencing of human early embryos. Nature cell biology20, 847-858, doi:10.1038/s41556-018-0123-2 (2018).
17 Petropoulos, S. et al. Single-Cell RNA-Seq Reveals Lineage and X Chromosome Dynamics in Human Preimplantation Embryos. Cell167, 285, doi:10.1016/j.cell.2016.08.009 (2016).
18 Wu, J. et al. Chromatin analysis in human early development reveals epigenetic transition during ZGA. Nature557, 256-260, doi:10.1038/s41586-018-0080-8 (2018).
19 Shahbazi, M. N. et al. Pluripotent state transitions coordinate morphogenesis in mouse and human embryos. Nature552, 239-243, doi:10.1038/nature24675 (2017).
20 Lyon, M. F. Gene action in the X-chromosome of the mouse (Mus musculus L.). Nature190, 372-373 (1961).
21 Qiu, W. et al. Spatio-temporal expression of matrix metalloproteinase-26 in human placental trophoblasts and fetal red cells during normal placentation. Biology of reproduction72, 954-959, doi:10.1095/biolreprod.104.036772 (2005).
22 Camolotto, S. et al. Expression and transcriptional regulation of individual pregnancy-specific glycoprotein genes in differentiating trophoblast cells. Placenta31, 312-319, doi:10.1016/j.placenta.2010.01.004 (2010).
23 Yadgary, L., Wong, E. A. & Uni, Z. Temporal transcriptome analysis of the chicken embryo yolk sac. BMC genomics15, 690, doi:10.1186/1471-2164-15-690 (2014).
24 Gerovska, D. & Arauzo-Bravo, M. J. Does mouse embryo primordial germ cell activation start before implantation as suggested by single-cell transcriptomics dynamics? Molecular human reproduction22, 208-225, doi:10.1093/molehr/gav072 (2016).


